Por Epidemiologos Argentinos Metadisciplinarios
A partir de datos obtenidos en Wuhan, República Popular China, el 11 de marzo de 2020, la Organización Mundial de la Salud (OMS) declaró una pandemia, basada en el aumento de casos de una enfermedad respiratoria de sintomatología variable, inespecífica y vagamente descripta como enfermedades anteriormente reconocidas, como un resfrío o gripe, hasta la clásica complicación con neumonía, especialmente en pacientes añosos e inmunodeprimidos, presuntamente asociada con un “nuevo” virus, hoy llamado SARS-CoV2. El supuesto agente causal fue apresuradamente caracterizado como un betacoronavirus envuelto, cuyo genoma se describe como ARN simple, de sentido positivo, compuesto por unas 30.000 bases, en un estudio carente de valor científico (Zhu, Zhang, Wang, & Li, 2020). Codifica para enzimas de replicación y proteínas estructurales, llamadas S (espícula), E (envoltura), M (membrana) y N (nucleocápside). Para cumplir su ciclo replicativo, se ha propuesto que la proteína S del virus haría contacto con una enzima de membrana citoplasmática, la ECA2 (enzima convertidora de angiotensina 2), que oficiaría de receptor, proporcionándole la entrada a la célula eucariota. Esta enzima se expresa mayoritariamente en testículo, riñón y corazón, y no en vías respiratorias, razón por la cual es altamente improbable que un virus desarrolle una enfermedad en un tejido donde no se expresa su receptor.
Los coronavirus son conocidos desde hace unos años, algunos por producir resfríos, otros son benignos del viroma humano (Popgeorgiev, Temmam, Raoult, & Desnues, 2013). También se conocen dos coronavirus, supuestamente zoonóticos, que también producen síndromes respiratorios: MERS-CoV (2012) y SARS-CoV (2002, 2003), éste último con asombrosa homología genética con este presunto SARS-CoV2.
Sin ninguna evidencia científica, y contra toda lógica, se pregona que MERS-CoV y SARS-CoV, han desaparecido de la faz de la Tierra.
PCR EN TIEMPO REAL, APLICADA A SARS-COV2
Es un método de laboratorio, que se basa en la detección cualitativa de una secuencia de material genético determinada, una pequeña porción de unos 200 nucleótidos, supuestamente pertenecientes al novel coronavirus SARS-CoV2.
El personal técnico que ejecuta esta prueba, desde la toma de muestra hasta la emisión del protocolo de informe, debe estar altamente capacitado.
El primer paso crítico es la correcta toma de muestra, que no debe estar contaminada por agentes ajenos a ella.
En el laboratorio, se hace una extracción del material genético presente en la muestra a analizar. Éste es un paso crucial, que emplea diferentes metodologías y marcas comerciales.
El proceso de PCR se lleva a cabo en un equipo llamado ciclador térmico, que está conectado a un software de doble interfaz, del que recibe las instrucciones de proceso (temperaturas y duración de cada etapa de la reacción) y hacia el cual envía los datos de la señal fluorescente detectada.
Dado que se trata de identificar un segmento de ARN (ácido ribonucleico) de cadena simple, el primer paso de esta técnica consiste en retro-transcribirlo a ADNc (ácido desoxirribonucleico complementario), por medio de una enzima, la transcriptasa reversa.
Luego se ejecuta la fase de amplificación (síntesis de millones de copias) de la secuencia buscada, en el termo-ciclador, acoplado a un detector óptico, que va midiendo fluorescencia a medida que se generan los amplicones (productos de amplificación de la reacción).
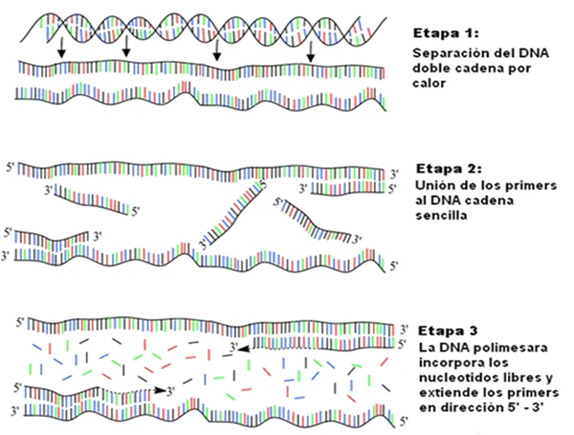
La reacción de PCR se produce dentro del equipo, en sucesivos ciclos, en los que se someten los pocillos de reacción a diferentes temperaturas, que permiten desnaturalizar el ADNc, exponer la secuencia diana que se desea detectar y amplificarla. Para esto se utilizan dos oligonucleótidos de diseño, llamados cebadores (primers), que se unen por complementariedad de bases nucleotìdicas a la secuencia de interés e indican el pequeño intervalo de nucleótidos sobre el ADNc, sobre el cual hibridiza también otro reactivo llamado sonda. Esta consiste, al igual que los cebadores, en una secuencia nucleotídica, diseñada ad hoc, que porta un fluoróforo inactivo (molécula capaz de generar señal fluorescente antes cambios químicos estructurales). La ADN polimerasa, es la enzima que irá copiando esos segmentos en los sucesivos ciclos, generando una amplificación exponencial de la secuencia diana buscada. Al generarse los amplicones, se activa la señal fluorescente, que es registrada por el detector, a medida que van transcurriendo los sucesivos ciclos de amplificación.
Amplificación exponencial:
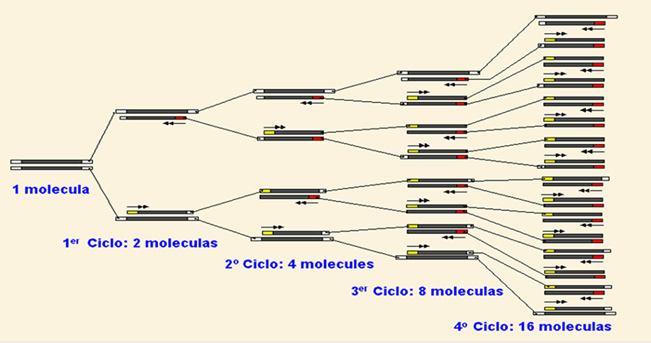
1er ciclo: 21 = 2 copias 2do ciclo: 22 = 4 copias
3er ciclo: 23 = 8 copias 4to ciclo: 24 = 16 copias
35to ciclo: 235 = 34 billones de copias!!!
Al finalizar todo el proceso, el software del equipo genera un gráfico integrando el comportamiento de las señales fluorescentes de cada muestra, versus el número de ciclos.
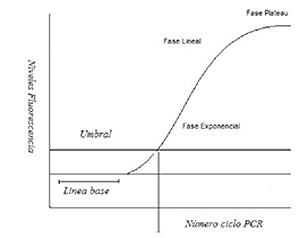
El producto de amplificación se detecta en tiempo real a medida que transcurren los ciclos térmicos, de tal manera que el nivel de fluorescencia registrada (ΔRn) aumenta exponencialmente. El Ct (cycle threshold = ciclo umbral) es el número de ciclos en el que la señal de fluorescencia de una determinada muestra cruza el umbral. Cuanto menor es el valor de Ct, mayor es la concentración inicial de la secuencia genética presente en la muestra.
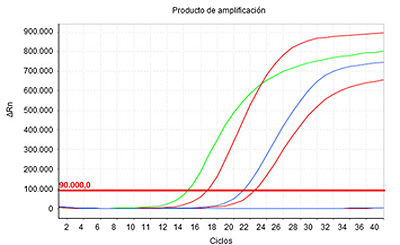
En el presente gráfico, se observan 4 muestras (curvas sigmoideas, de crecimiento exponencial, de distintos colores) que han superado el umbral preestablecido, en diferentes ciclos de amplificación.
Por ejemplo, la muestra que corresponde a la línea verde, alcanzó el umbral en el ciclo 14 (Ct 14), la azul en el 21 (Ct 21).
Este es un punto crítico, donde se debe adoptar un criterio consensuado, en base a la validación de la PCR contra el cultivo viral en líneas celulares, con el objetivo de estandarizar el número de ciclos que se considere adecuado como punto de corte para considerar una muestra como positiva o negativa y permita inferir de manera más precisa infección e infectividad. En el ámbito clínico, la mera detección de pequeñas secuencias genéticas, atribuidas a un virus no tienen ninguna utilidad para el Diagnóstico, si el resultado no se contrasta con clínica, epidemiología y otros estudios complementarios (Rx, TAC, hemograma, etc)
En principio, la OMS y la OPS (Organización Panamericana de la Salud) recomendaron un protocolo de rT-PCR (Corman, y otros, 2020), creado por el equipo del doctor Christian Drosten, del Instituto de Virología de la Universidad de Berlín, Hospital Charité, publicado el 23 de enero de 2020.
El Centro Nacional de Referencia para la Vigilancia de Virus Respiratorios del Instituto Carlos G. Malbràn, adoptó para toda la red de vigilancia este protocolo, estableciendo inicialmente el siguiente criterio para definir muestras como positivas o negativas:
Hasta Ct 37: positivo (genoma detectable)
Entre Ct 37 y 40: no conclusivo, repetir la reacción con doble volumen de muestra extraída. Si el Ct no cambia, se considera como No detectable (resultado negativo). Si el Ct disminuye, se informa Detectable (positivo).
Es necesario aclarar que estos puntos de cortes fueron establecidos para la detección del gen E, común para todos los betacoronavirus del grupo B (Género Sarbecovirus). Si bien el protocolo del Charitè propone una segunda PCR para detección de gen RdRp, (utilizando un sistema de sondas que permitiría diferenciar al novel coronavirus SARS CoV-2 de los otros sarbecovirus, como el SARS del 2002) en la actualidad no se está haciendo la determinación de gen RdRp, debido a que tiene menor sensibilidad y porque el Centro de Referencia asume que el SARS CoV-1 no estaría circulando.
CONSIDERACIONES
- El test tiene altísima sensibilidad analítica, pero la presencia de ARN viral no implica presencia de viriones infectivos. La clínica es soberana. El profesional médico debe evaluar el diagnóstico clínico de una enfermedad, reuniendo toda la información y una serie de criterios preestablecidos, revisados y consolidados. La RT-PCR no se considera una prueba diagnóstica en sí misma de infección viral sin un contexto, ya que sólo detecta un fragmento de material genético, que podría corresponderse o no con un virus, siendo éste infectivo o no (Jefferson, Spencer, Brassey, & Heneghan, 2020).
- El resultado de este test no puede ser confirmado por otro procedimiento diagnóstico. Los métodos de laboratorio de screening, de alta sensibilidad como éste, detectan gran cantidad de falsos positivos, que luego deben ser confirmados por pruebas más específicas.
- La prueba RT-PCR nunca fue validada contra un gold standard (cultivos virales), sólo se aceptócomo un dogma, con la excusa de una supuesta emergencia sanitaria. Cabe aclarar que los cultivos virales hechos hasta el momento sólo analizan citopatología, es decir, observan daños en las células cultivadas, los cuales pueden deberse a múltiples causas.
- No se han comprobado los postulados de Koch adaptados a virus, en cuanto al agente etiológico de covid-19, es decir, correlación virus microfotografiado con material genético viral y con la patología que se le atribuye.
- El inserto de la prueba RT-PCR, sólo para uso de emergencia, de los Centros de Prevención y Control de Enfermedades de Estados Unidos (CDC), en su revisión del 13 de julio de 2020 (CDC 2019-Novel Coronavirus (2019-nCoV) Real-Time RT-PCR Diagnostic Panel, For Emergency Use Only) señala en su página 39, bajo el título “Analytical Performance” “Limit of detection (LoD)”: “…dado que no existen virus aislados cuantificados disponibles en la actualidad…”. Por otro lado, en la publicación de Drosten también señalan que para determinar la sensibilidad de la técnica, utilizaron viriones de SARS CoV (no de SARS CoV 2) obtenidos de cultivos de células Vero y para estudiar el límite de detección (LoD) utilizaron RNA sintético, transcripto in vitro. Es decir, no se cuenta con virus aislados cuantificados.
- Los reactivos de RT-PCR para detectar SARS-CoV2 no están estandarizados. Se han fabricado y aplicado masivamente para diagnóstico de covid-19 durante la pandemia en múltiples versiones de diferentes marcas comerciales, cada una de las cuales diseña sus propios cebadores y sondas y determina las regiones genéticas hacia los cuales van dirigidos. Los fabricantes de kits comerciales no informan las secuencias de los primers y sondas que utilizan, con lo cual es imposible evaluar la posibilidad de reacciones cruzadas con otras secuencias de diferente origen, ni evaluar la especificidad del test.
- Las pruebas de RT-PCR no son específicas para SARS CoV-2. Los test están diseñados para detectar también otros coronavirus del tipo SARS del Genero Sarbecovirus (incluyendo al SARS CoV-1 referido como el agente del brote del año 2002-2003 y otros virus de murciélagos asiáticos).
- El umbral de ciclos para informar un resultado como positivo o negativo no está estandarizado. Según una revisión de la Universidad de Oxford (Jefferson, Spencer, Brassey, & Heneghan, 2020) estudios reportaron ausencia de crecimiento viral en cultivos celulares en base al valor de corte (Ct) de la RT-PCR. Los valores de Ct a partir de los cuales no se obtiene crecimiento viral comprenden un rango desde Ct >24 hasta > 34.
- Del punto anterior, y según los autores del estudio (Jefferson, Spencer, Brassey, & Heneghan, 2020), se estarían obteniendo gran número de falsos positivos de la prueba RT-PCR, segregando personas por ser clasificadas como infectivas, siendo sometidas a discriminación y confinamiento forzado, a pesar de no representar una amenaza para la salud pública.
- Las pruebas PCR carecen de especificidad clínica para detectar covid-19, dado que arrojan hasta un 80 % de resultados falsos positivos. Un estudio, publicado el 5 de marzo de 2020, estudió falsos positivos de contactos estrechos, detectando 80,33% de falsos positivos (Zhuang, y otros, 2020). Este estudio, cuyo autor principal tiene publicados más de 1700 trabajos científicos, sorpresivamente fue retirado de la Chinese Medical Association Publishing House Ltd., sin embargo, el abstract quedó publicado en PubMed. Según el boletín epidemiológico oficial de la Ciudad Autónoma de Buenos Aires, Argentina, de julio de 2020, había en ese momento 38% de asintomáticos, cifra que se extendió al 50% en el siguiente boletín.
- El test PCR carece de valor predictivo positivo para el Síndrome COVID-19. Se demostró que el valor predictivo positivo (capacidad de discernir si un individuo está enfermo cuando la prueba da positiva) es de sólo 19,67%. (Zhuang, y otros, 2020). En términos epidemiológicos, el valor predictivo positivo mide la eficacia real de una prueba diagnóstica.
BIBLIOGRAFÍA
Corman, V., Landt, O., Kaiser, M., Molenkamp, R., Meijer, A., Chu, D., y otros. (2020). Detection of 2019 novel coronavirus (2019-nCoV) by real-time RT-PCR. Euro Surveillance, 3(25), 2000045.
Jefferson, T., Spencer, E., Brassey, J., & Heneghan, C. (2020). Viral cultures for COVID-19 infectivity assessment. Systematic review. MedRXIV, 10-20.
Popgeorgiev, N., Temmam, S., Raoult, D., & Desnues, C. (2013). Describing the silent human virome with an emphasis on giant viruses. Intervirology, 56(6), 395-412.
Zhu, N., Zhang, D., Wang, W., & Li, X. (24 de Enero de 2020). A novel coronavirus from patients with pneumonia in China. New English Journal of Medicine(382), 727-733.
Zhuang, G., Shen, M., Zeng, L., Mi, B., Chen, F., Liu, W. P., y otros. (2020). Potential false-positive rate among the ‘asymptomatic infected individuals’ in close contacts of COVID-19 patients. Chinese Medical Association Publishing House Ltd., 41(4), 485-488.
Fuente:
Comisión PCR / Epidemiologos Argentinos Metadisciplinarios, vía Ciencia y Salud Natural — Reacción en cadena de la polimerasa en tiempo real con retrotranscripción (RT-PCR), para la detección de sars-cov-2.